La révolution du très haut débit
Le développement depuis 2009 des technologies de séquençage de nouvelle génération (NGS, Next Generation Sequencing) constitue une révolution technologique sans précédent. Alors que le séquençage d’un seul génome humain a nécessité 10 ans de collaborations internationales (1993-2003) et 2,7 milliards de dollars, il est désormais possible de séquencer l'exome humain en l’espace de quelques semaines, voire de quelques jours, pour un coût inférieur à 1 000 euros. Le séquençage de génomes humains entiers, composé de 3 milliards de paires de bases, a déjà été réalisé à visée médicale par quelques équipes aux États-Unis, au Royaume-Uni, aux Pays-Bas et en France. Grâce notamment au multiplexage (séquençage de plusieurs échantillons, identifiés par code-barre, au sein d'un même run), il est possible d'augmenter les capacités de séquençage et d'en diminuer le coût. Ces avantages sont essentiels à la mise en place d'un diagnostic génétique étendu au plus grand nombre de patients. Parallèlement, les progrès en matière de techniques de gestion et d'analyse des données massives (Big Data) et de nouvelles infrastructures de stockage doivent permettre d'absorber cette augmentation des capacités de séquençage afin de prendre en charge des données de plus en plus nombreuses.
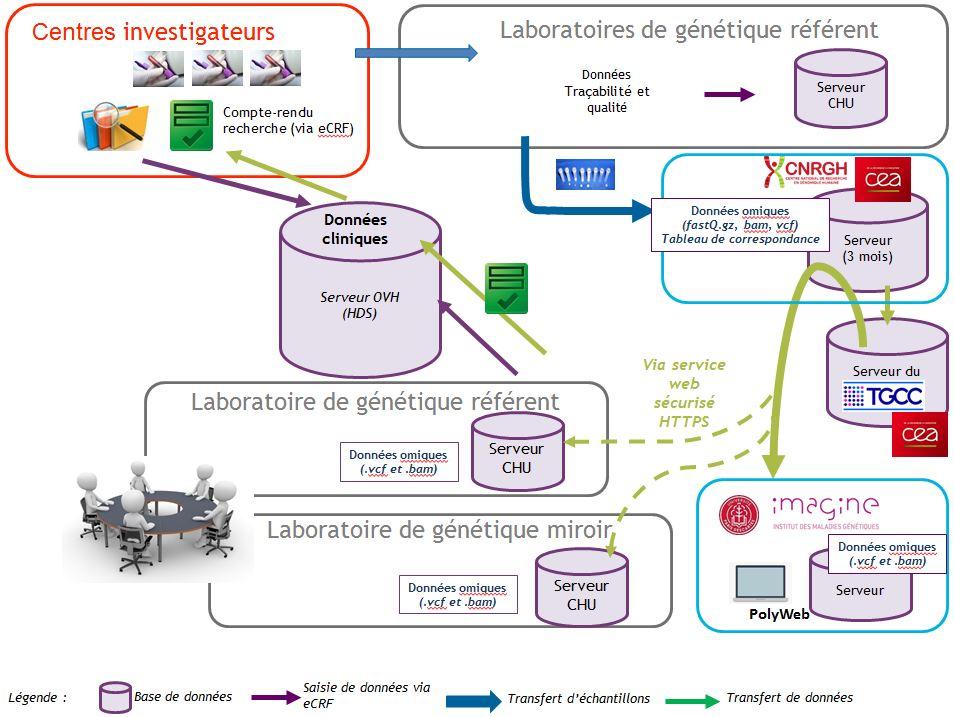
Parcours génomique des échantillons DEFIDIAG
Les échantillons biologiques de l’étude principale (sanguins) sont envoyés au laboratoire de génétique DEFIDIAG (laboratoire référent) auquel le centre d’investigation est affilié qui réalise l’extraction des acides nucléiques (ADN) à partir des échantillons. Les échantillons d’acides nucléiques sont ensuite envoyés au Centre National de Recherche en Génomique Humaine (CNRGH, Evry) qui réalise le séquençage génétique de ces échantillons.
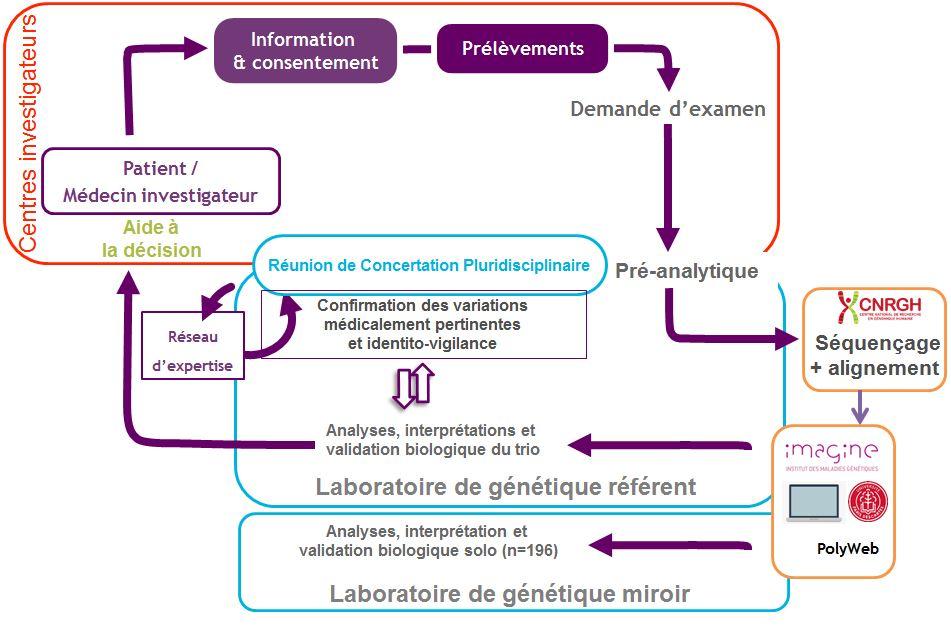
Parcours des données du génome
Les données générées suite au séquençage des échantillons sont transférées du CNRGH à la plateforme de bio-informatique de l’Institut Imagine qui met à disposition l’outil de visualisation des données (PolyWeb). Les données sont alors disponibles pour visualisation et interprétation par les personnels habilités des laboratoires de génétique DEFIDIAG (laboratoire référent pour les données de génome du trio et laboratoire miroir pour les données du cas index) qui poursuivent l’analyse des données génétiques. Une fois tous les 15 jours, une Réunion de Concertation Pluridisciplinaire (RCP) regroupant les laboratoires de génétique DEFIDIAG deux par deux rassemblant un comité composé notamment de médecins, de bio-informaticiens et de biologistes moléculaires permet d’aboutir à un résultat d’analyse du génome . Les professionnels gardent donc un rôle essentiel dans l’interprétation de ces données génétiques. Le laboratoire de génétique rédige alors un compte-rendu biologique qui est envoyé au médecin investigateur du centre ayant réalisé l’inclusion du trio qui à son tour annoncera les résultats au trio. En cas de mise en évidence d'une variation génétique ou d'une anomalie chromosomique en cause dans la maladie du cas index et en cas de risque de transmission lors d’une future grossesse pour un autre enfant, ces analyses pourront donner accès à un diagnostic prénatal ou au diagnostic préimplantatoire.